 scGPT toward building a foundation model for single-cell multi-omics using generative AI
scGPT toward building a foundation model for single-cell multi-omics using generative AI
题目: scGPT:利用生成式人工智能构建单细胞多组学的基础模型
DOI: https://doi.org/10.1038/s41592-024-02201-0 (opens new window)
Cite:Cui, H., Wang, C., Maan, H. et al. scGPT: toward building a foundation model for single-cell multi-omics using generative AI. Nat Methods (2024).
Pubmed:https://www.nature.com/articles/s41592-024-02201-0/ (opens new window)
作者介绍:
| Bo Wang |
|---|
 |
| Peter Munk Cardiac Centre, University Health Network, Toronto, Ontartio, Canada |
| bowang@vectorinstitute.ai |
# Abstract:
生成式预训练模型在语言和计算机视觉等各个领域取得了显著成功。特别是结合大规模多样化的数据集和预训练的 transformers,已经成为开发基础模型的有前途的方法。通过语言和细胞生物学之间的类比(文本由单词组成;同样,细胞由基因定义),我们的研究探讨了基础模型在推进细胞生物学和遗传学研究中的适用性。利用快速增长的单细胞测序数据,我们构建了一个用于单细胞生物学的基础模型,scGPT,这个模型基于一个跨越超过3300万个细胞的生成式预训练 transformers。我们的研究结果表明,scGPT能够有效地提取有关基因和细胞的重要生物学见解。通过进一步的迁移学习适应,scGPT可以优化以在各种下游应用中实现卓越性能。这些应用包括细胞类型注释、多批次整合、多组学整合、干扰响应预测和基因网络推断等任务。
# Results:
# Figure 1: Single-cell transformer foundation model overview.
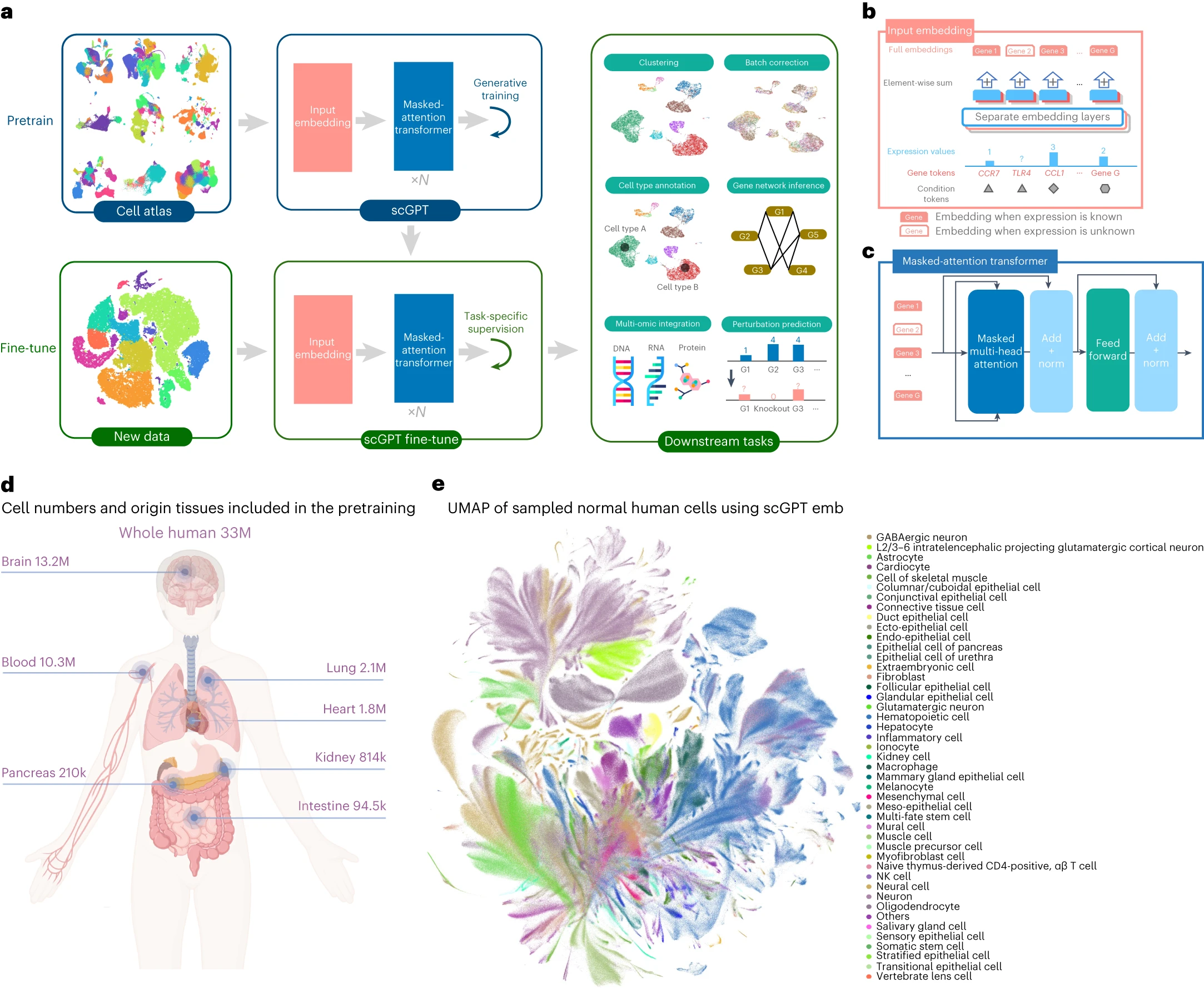
Figure 1. Single-cell transformer 基础模型概述
(a) scGPT 的工作流程。该模型在来自细胞图谱的大规模 scRNA-seq 数据上进行生成性预训练。scGPT 的核心组件包含堆叠的 Transformer 块,带有用于生成训练的专门注意力掩码。对于下游应用,可以在新数据上微调预训练的模型参数。我们将 scGPT 应用于多种任务,包括细胞类型注释、批次校正、多组学整合、遗传扰动预测和基因网络推断。
(b) 输入数据嵌入的详细视图。输入包含三层信息:基因标记、表达值和条件标记(模态、批次、扰动条件等)。
(c) scGPT Transformer 层的详细视图。我们在掩码多头注意力块中引入了专门设计的注意力掩码,以对单细胞测序数据进行生成性预训练。Norm 表示层规范化操作。
(d) 说明训练数据的大小和来源器官的图表。 scGPT 全人类模型在 3300 万 (M) 个正常人类细胞的 scRNA-seq 数据上进行了预训练。
(e) 预训练的 scGPT 细胞的 UMAP 可视化。
# Figure 2: Cell type-annotation results using scGPT.
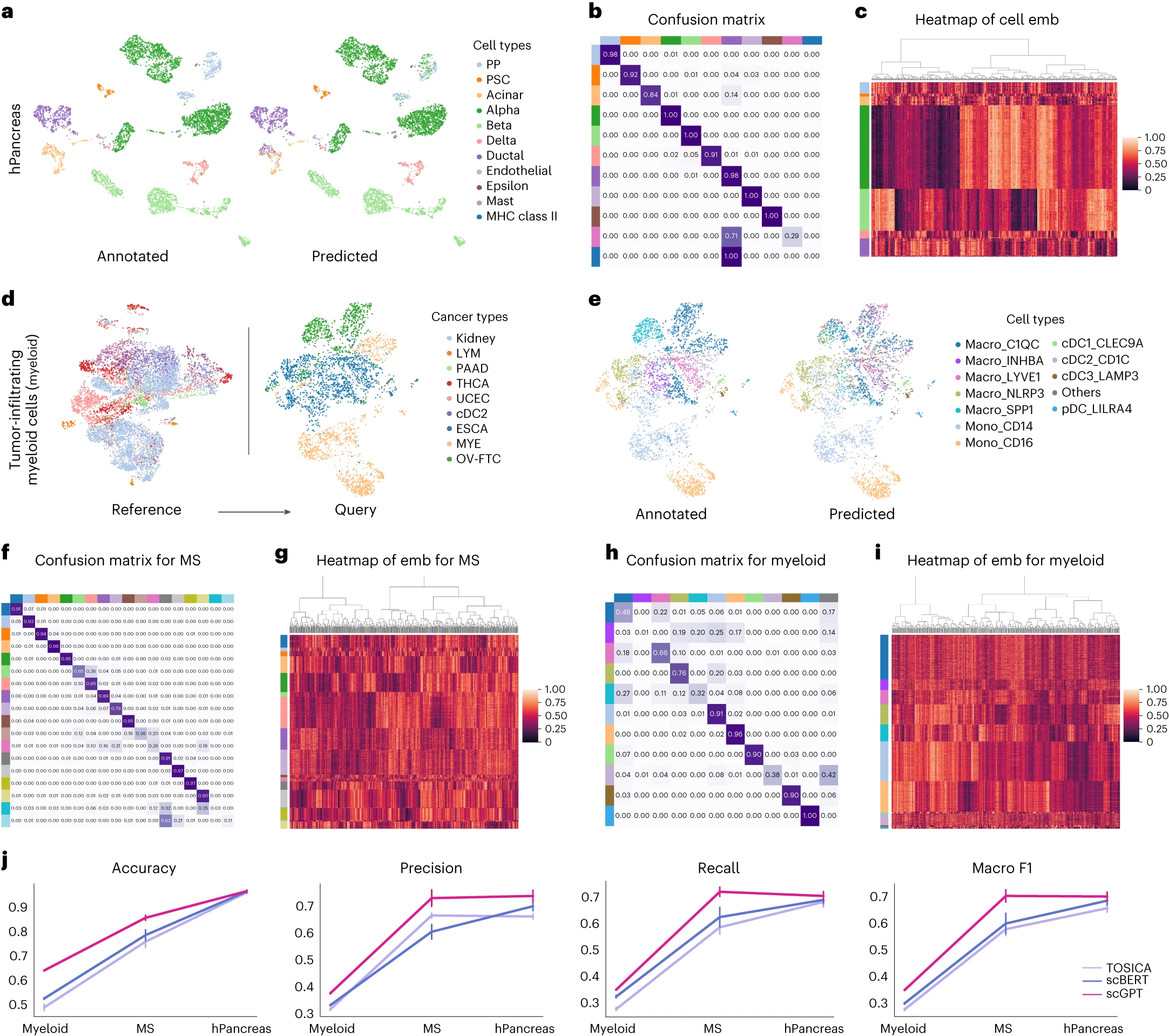
Figure 2. 使用 scGPT 的细胞类型注释结果
(a) 来自人类胰腺数据集的细胞基因表达的 UMAP,按原始研究中注释的细胞类型(左)和微调的 scGPT 预测的细胞类型(右)着色。
(b) 人类胰腺数据集中预测和注释的细胞类型之间的混淆矩阵。
(c) 人类胰腺数据集中来自 scGPT 的 512 维细胞嵌入的热图。
(d) 髓系数据集的 UMAP 可视化,按癌症类型着色。scGPT 在参考分区(左)上进行了微调,并在查询分区(右)上进行了评估。
(e) 在查询分区中,UMAP 按原始研究中注释的细胞类型(左)和 scGPT 预测的细胞类型着色。
(f,h) 分别为 MS 和髓系数据集的预测细胞类型和实际注释之间的混淆矩阵。
(g,i) 分别显示 MS 和髓系数据集中细胞的 scGPT 512 维细胞嵌入的热图。
(j) 通过对髓系、MS 和人类胰腺数据集进行n = 5 次随机训练-验证分割来评估 scGPT 的细胞注释性能。
# Figure 3: Prediction results for perturbation response and reverse perturbation.
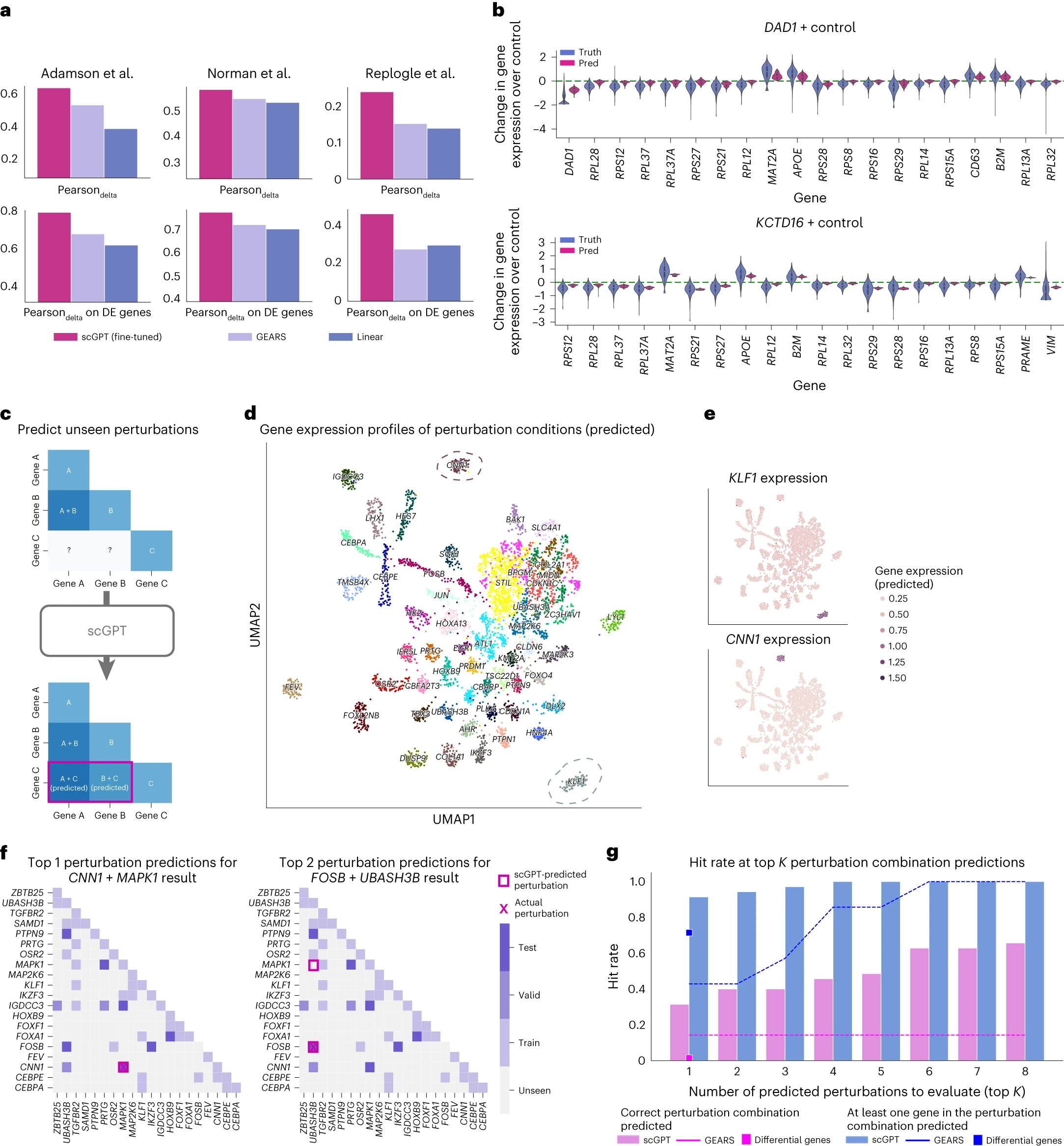
Figure 3. 扰动响应和逆扰动的预测结果
(a) scGPT 与其他扰动预测方法的比较。报告了预测基因表达变化和实际基因表达变化之间的皮尔逊相关性。
(b) Adamson 测试数据集中的两个示例扰动,前 20 个差异表达基因的预测和实际基因表达变化的分布。
(c) 使用 scGPT 预测看不见的扰动响应的示意图。
(d) 扰动条件下预测的基因表达谱的 UMAP。
(e) 两个选定的扰动基因(KLF1和CNN1)在扰动条件的 UMAP 上的表达模式。
(f) 在 20 个基因的扰动组合空间中可视化可能的扰动组合。
(g) 与 GEARS 和前两个差异基因的幼稚基线相比,scGPT 在七个测试用例中正确和相关预测的前 1-8 名准确率。
# Figure 4: Results of multi-batch and multi-omic integration.

Figure 4. 多批次和多组学整合的结果
(a) 在 PBMC 10k 数据集上针对细胞类型聚类任务对微调的 scGPT 进行基准测试。
(b) 在 10x Multiome PBMC 数据集(配对 RNA 和转座酶可及染色质 (ATAC) 数据检测)上使用 scGLUE 13和 Seurat (v.4) 进行微调的 scGPT 模型的基准测试,用于细胞类型聚类任务。
(c) 在 BMMC 数据集(配对 RNA 和蛋白质数据)上使用 Seurat(v.4)对微调的 scGPT 模型进行细胞类型聚类任务的基准测试。
(d) 在 ASAP PBMC 数据集(镶嵌 RNA、ATAC 和蛋白质数据)上使用 scMoMat 对 scGPT 进行批次校正和细胞类型聚类任务的基准测试。
# Figure 5: Analysis of gene token embeddings.
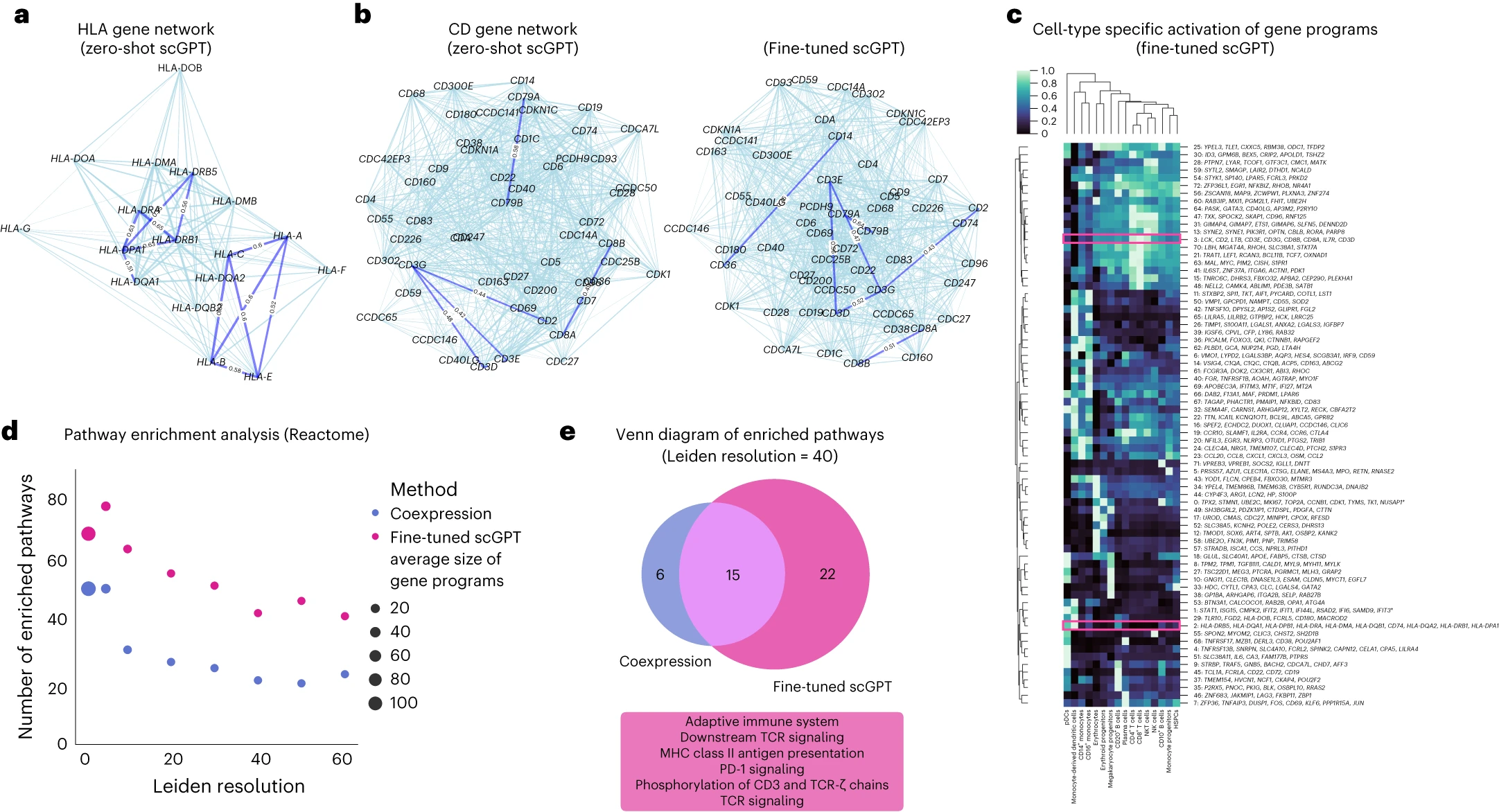
Figure 5. 基因标记嵌入的分析
(a) 来自零样本 scGPT 的 HLA 基因网络。
(b) 来自免疫人体数据集上零样本(即预训练)和微调 scGPT 的 CD 基因网络。
(c) 免疫人体数据集中scGPT提取的基因程序中的细胞类型特异性激活。颜色表示平均基因表达。对于包含超过 10 个基因的基因程序(用星号表示),为简单起见,仅显示前 10 个基因。
(d) 免疫人体数据集中 scGPT 提取的基因程序和共表达网络的通路富集分析。在不同莱顿分辨率下,将来自 scGPT 基因程序的富集通路数量与共表达方法的富集通路数量进行了比较。
(e) 维恩图比较了共表达和 scGPT 鉴定的富集通路之间的重叠和差异。文本框中突出显示了一些 scGPT 独有且特定于适应性免疫功能的示例通路。
# Figure 6: Attention-based gene interaction analysis.
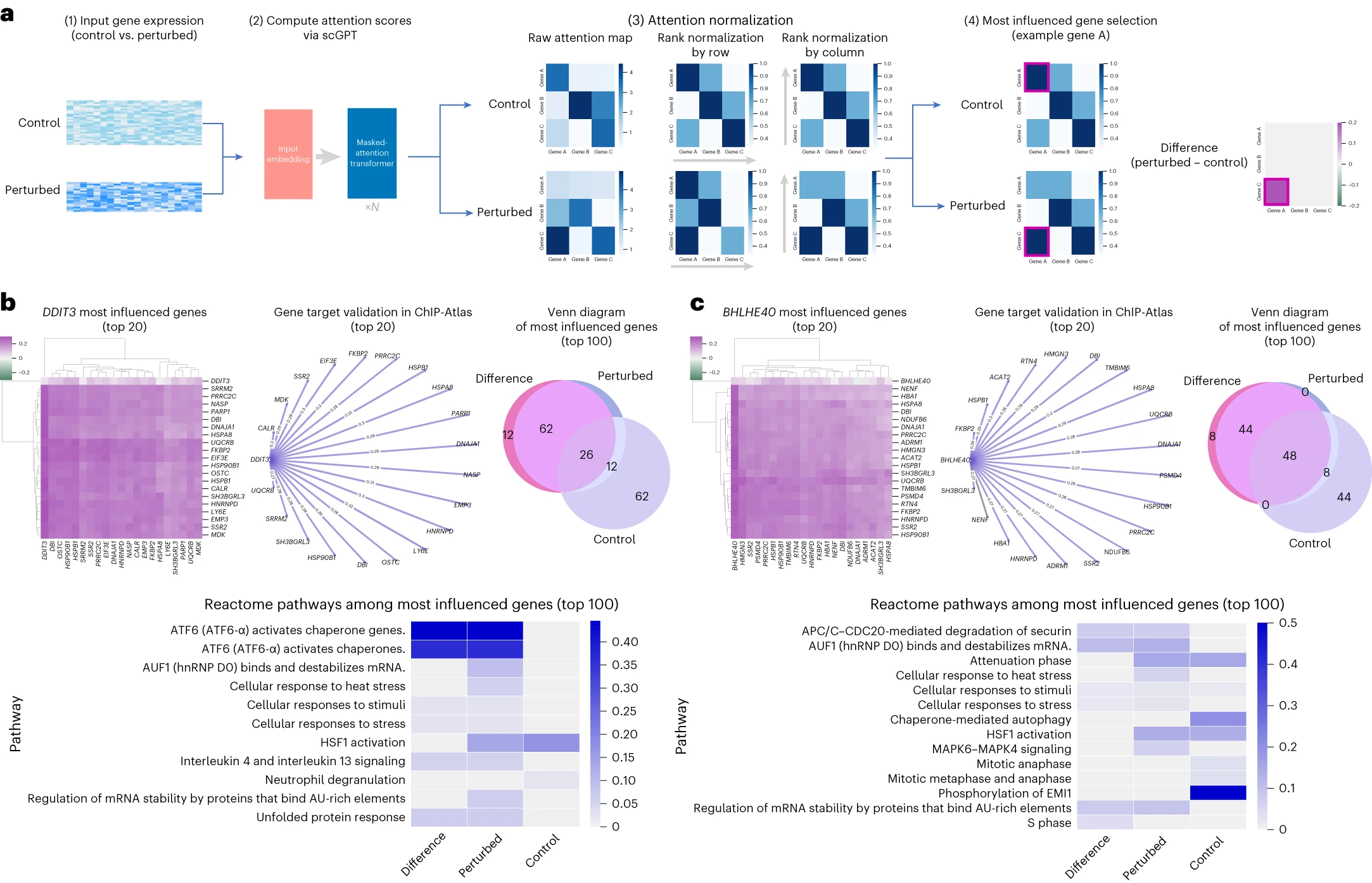
Figure 6. 基于注意力的基因相互作用分析
(a) 基于注意力的 GRN 发现扰动数据工作流程。
(b) DDIT3抑制的 GRN 分析。基因连接热图显示了受DDIT3抑制影响最大的前 20 个基因网络的扰动后变化。基因靶标网络图展示了在 ChIP-Atlas 数据库中验证的前 20 个基因,其中 ChIP-seq 预测的靶标以紫色突出显示。维恩图比较了在三种选择设置(即对照、扰动和扰动后差异)中确定的前 100 个最受影响的基因集之间的重叠和差异。通路热图展示了在这三种选择设置中从前 100 个最受影响的基因中确定的 Reactome 通路的差异。颜色表示每个通路由基因重叠百分比表示的强度。
(c) 对BHLHE40抑制的GRN分析,以类似的方式可视化。
# Discussion:
我们引入了 scGPT,这是一种基础模型,它利用预训练的 transformer 的强大功能处理大量单细胞数据。通过利用 transformer 的注意力机制,scGPT 可以在单细胞水平上捕获基因与基因之间的相互作用,从而提供额外的可解释性。我们通过零样本和微调设置中的全面实验展示了预训练的好处。对于未来的发展方向,我们计划在更大规模、更多样化的数据集上进行预训练,包括多组学数据、空间组学和各种疾病状况。
