 Spatial profiling of chromatin accessibility in mouse and human tissues
Spatial profiling of chromatin accessibility in mouse and human tissues
题目: 小鼠和人体组织中染色质可及性的空间分析
DOI: https://doi.org/10.1038/s41586-022-05094-1 (opens new window)
Cite: Deng, Y., Bartosovic, M., Ma, S. et al. Spatial profiling of chromatin accessibility in mouse and human tissues. Nature 609, 375–383 (2022).
作者介绍:
| Rong Fan |
|---|
 |
| Department of Biomedical Engineering, Yale University, New Haven, CT, USA |
| rong.fan@yale.edu |
# Abstract:
组织中的细胞功能取决于局部环境,需要新的方法来对组织环境中的生物分子和细胞进行空间绘图。空间转录组学的出现使得基因组规模的基因表达作图成为可能 ,但缺乏在细胞水平和基因组规模捕获组织空间表观遗传信息的能力。在这里,我们描述了一种使用新一代测序 (spatial-ATAC-seq) 结合原位 Tn5 转座化学 和微流控确定性条形码对组织切片进行空间分辨染色质可及性分析的方法。使用空间 ATAC-seq 对小鼠胚胎进行分析,描绘了组织区域特异性的表观遗传景观,并确定了参与中枢神经系统发育的基因调节因子。绘制小鼠和人类大脑中可访问的基因组图谱揭示了大脑区域的复杂区域化。将空间 ATAC-seq 应用于扁桃体组织解决了淋巴滤泡和滤泡外区域中免疫细胞类型和状态的空间独特组织。该技术通过实现空间解析的染色质可及性分析来促进空间生物学的发展,以提高我们对与发育和疾病的表观遗传基础相关的细胞身份、细胞状态和细胞命运决定的理解。
# Main:
单细胞测序提供了一种定义细胞类型和状态的切实方法,但组织解离过程会导致空间背景的丢失。此外,单细胞技术中的分离方法可能会由于解离或其他环境应激而优先选择某些细胞类型或扰乱细胞状态。空间转录组学的出现就是为了解决这些挑战,并改变我们在天然组织环境中描述细胞功能和状态的方式。为了研究组织环境中不同细胞类型和功能的空间组织的机制,非常需要不仅检查基因表达,还需要以空间解析的方式检查表观遗传基础,例如染色质可及性。空间表观遗传图谱将帮助我们揭示决定组织组织和功能驱动因素的因果关系。迄今为止,还缺乏直接在基因组规模和细胞水平的组织切片中空间绘制表观遗传状态(例如染色质可及性)的能力。
开发了使用测序 (ATAC-seq) 的转座酶可及染色质检测方法,并进一步应用于单细胞。使用 Tn5 (ATACsee)中组装的荧光标记 DNA 寡聚物对固定细胞中染色质可及性进行成像表明,原位分析染色质可及性是可行的。对特定区域的组织进行显微解剖,然后进行 scATAC-seq,可以对感兴趣区域的可接近染色质进行分析。然而,在细胞水平上对组织切片进行空间解析的染色质可及性绘图还不可能。在这里,我们对 DNA 寡聚物应用了空间条形码方案,这些寡聚物通过 Tn5 转座插入可访问的基因组位点,以实现空间 ATAC-seq:在细胞水平上对组织中染色质可访问性进行高空间分辨率全基因组图谱。小鼠胚胎的结果描绘了参与中枢神经系统(CNS)发育的区域特异性表观遗传景观和基因调节因子。我们还将空间表观基因组学应用于人体组织,包括扁桃体和海马体。Spatial-ATAC-seq揭示了与淋巴滤泡和滤泡外区域相关的免疫细胞类型和状态的空间独特组织。
# Results:
# Figure 1: Spatial-ATAC-seq design, workflow and data quality.
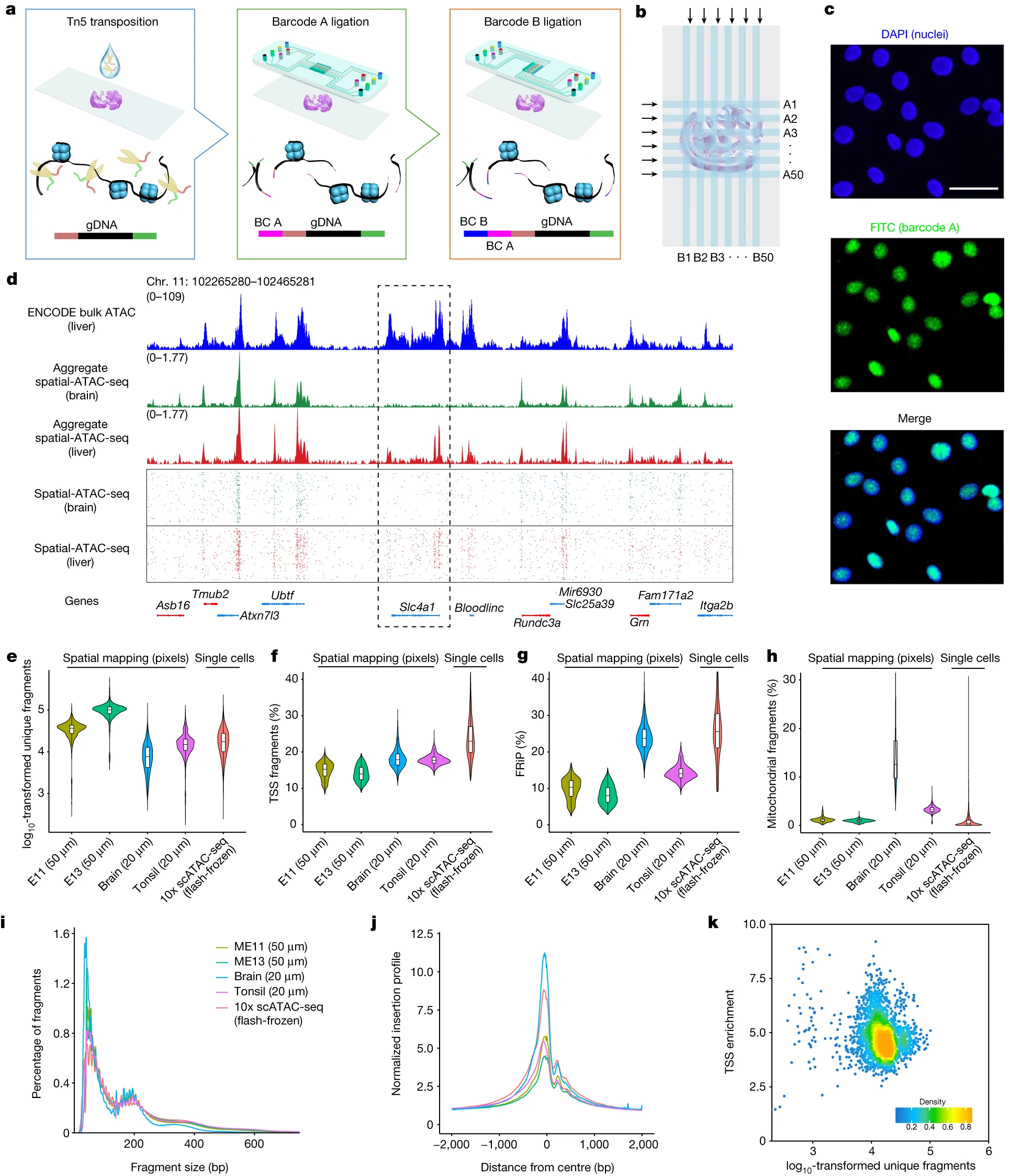
Figure 1. Spatial-ATAC-seq 设计、工作流程和数据质量
(a) 工作流程示意图。在组织切片中进行 Tn5 转座,然后原位连接两组 DNA 条形码(A1–A50、B1–B50)。
(b) 微流体横流方案。
(c) 使用荧光DNA探针验证原位转座和连接。Tn5 转座在 DAPI(蓝色)染色的载玻片上的 3T3 细胞中进行。接下来,将 FITC 标记的条形码 A 连接至转座酶可访问的基因组 DNA 上的接头。
(d) 聚合空间染色质可及性概况概括了已发表的 E13 小鼠胚胎肝脏中 ATAC-seq 的概况。
(e) spatial-ATAC-seq 和 10x scATAC-seq 之间独特片段数量的比较。
(f) spatial-ATAC-seq 和 10x scATAC-seq 之间 TSS 片段比例的比较。
(g) spatial-ATAC-seq 和 10x scATAC-seq 之间线粒体片段分数的比较。
(h) spatial-ATAC-seq 和 10x scATAC-seq 之间的峰读取分数 (FRiP) 的比较。
(i) spatial-ATAC-seq 和 10x scATAC-seq 之间 ATAC-seq 片段插入大小分布的比较。
(j) spatial-ATAC-seq 和 10x scATAC-seq 之间 TSS 周围 ATAC-seq 读段的富集度比较。
(k) TSS 富集分数与人扁桃体中每个细胞的独特核片段的比较。
# Figure 2: Spatial chromatin accessibility mapping of E13 mouse embryos.
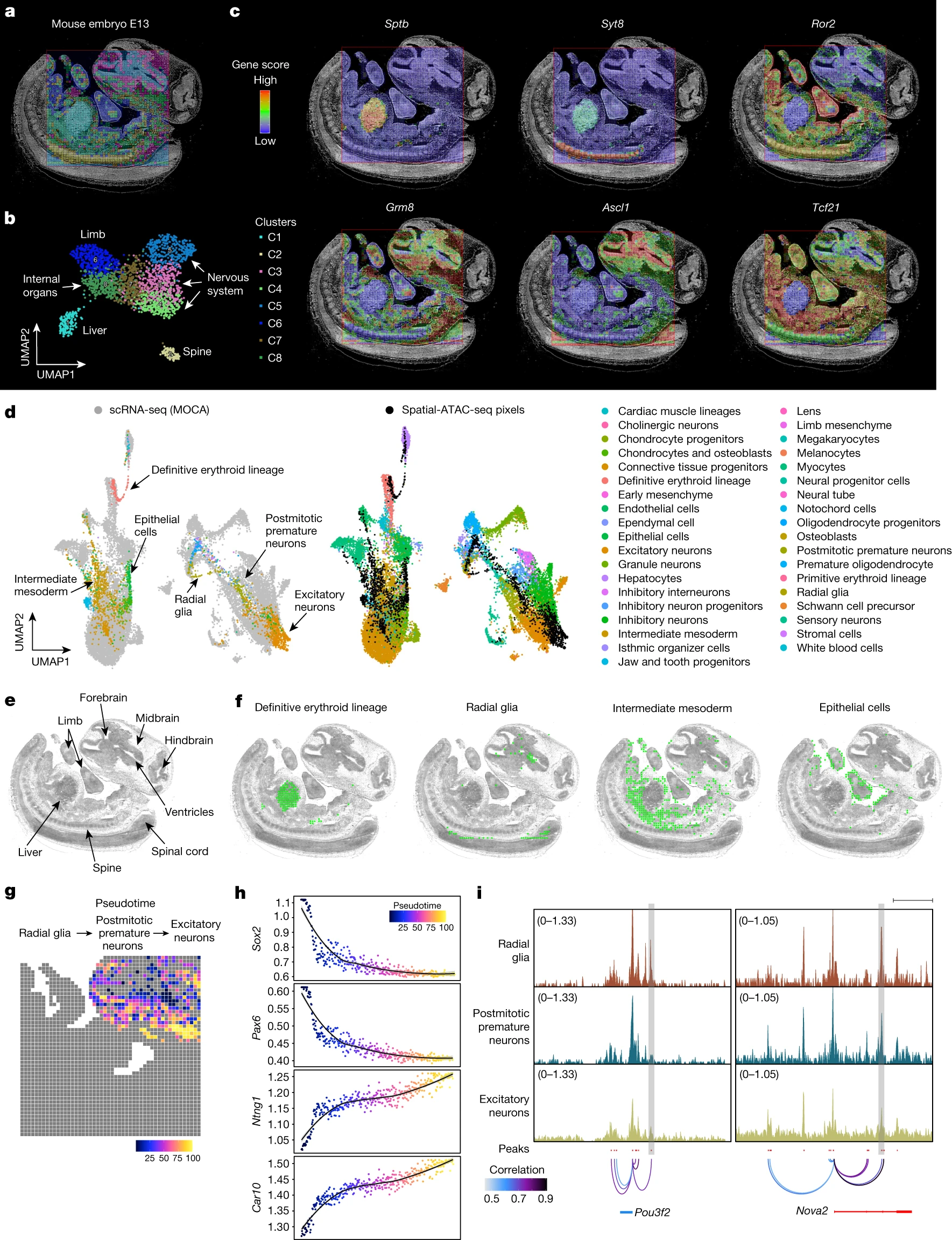
Figure 2. E13 小鼠胚胎的空间染色质可达性图谱
(a) 根据所有组织像素(50 μm 像素大小)的染色质可及性进行无偏聚类分析。簇与组织图像的叠加表明,空间染色质可及性簇与解剖区域精确匹配。为了更好的可视化,我们缩放了像素的大小。
(b) 用于染色质可及性的无监督聚类分析的 UMAP 嵌入。
(c) 不同簇中选定标记基因的基因得分的空间映射和选定基因的染色质可及性具有高度组织特异性。
(d) 来自 E13.5 小鼠胚胎的 scRNA-seq 整合和空间 ATAC-seq 数据。组合数据的无监督聚类按不同的细胞类型着色。
(e) 基于苏木精和伊红 (H&E) 染色图像的主要组织区域的解剖注释。
(f) 通过从 scRNA-seq 转移到空间 ATAC-seq 数据的标签识别的选定细胞类型的空间映射。
(g) 从放射状胶质细胞、有丝分裂后的早产神经元到绘制在空间中的兴奋性神经元的发育过程的伪时间重建。
(h) 所选基因的基因分数沿g中所示的伪时间的动态。
(i) Pou3f2和Nova2各个调控元件的染色质可及性动态(以灰色框突出显示)。
# Figure 3: Spatial chromatin accessibility mapping and integrative analysis of P21 mouse brain with a 20 µm pixel size.
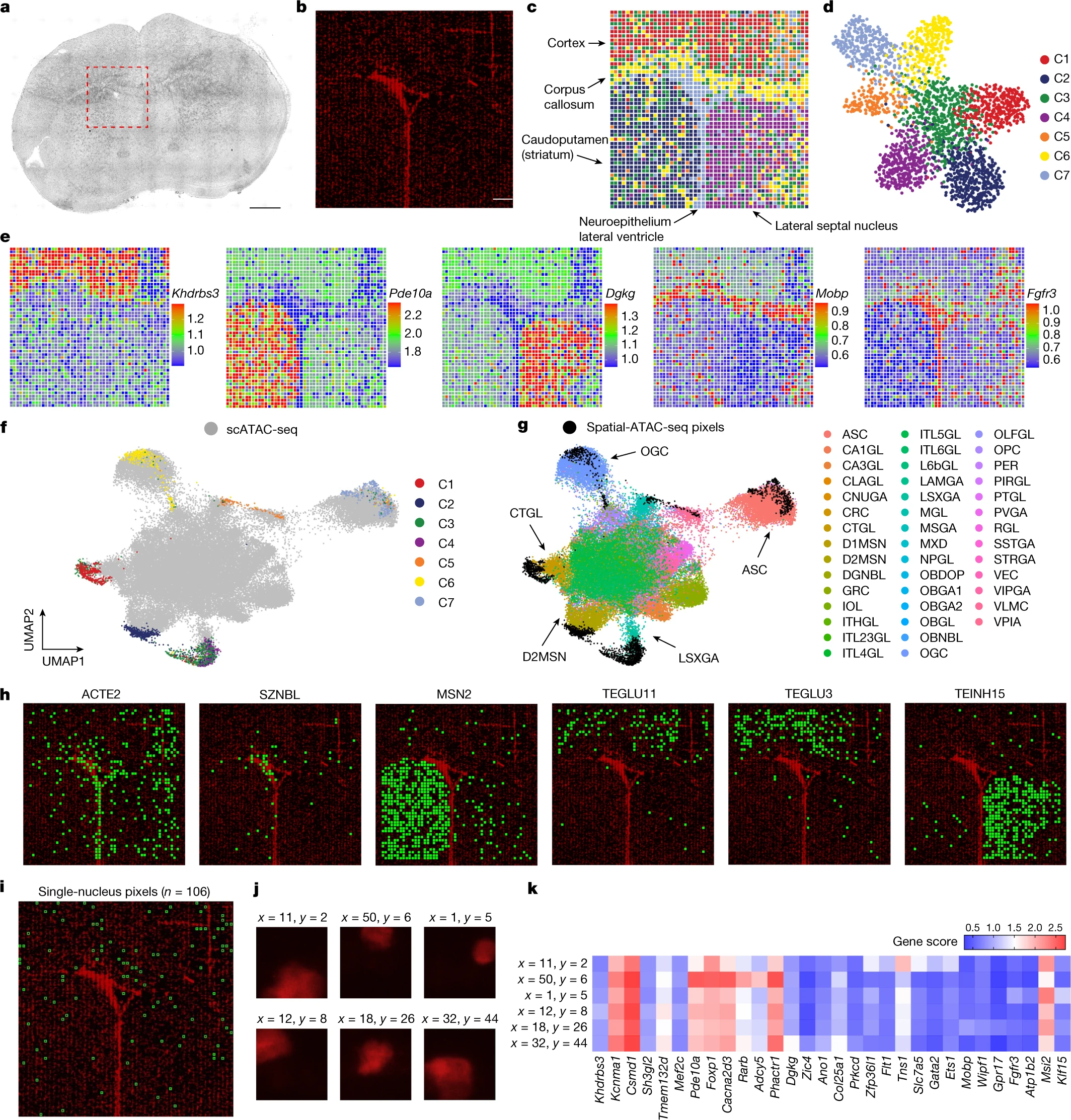
Figure 3. 20 µm 像素大小的 P21 小鼠大脑的空间染色质可及性图谱和综合分析
(a) 小鼠脑组织切片和感兴趣区域的明场图像(红色虚线框)。
(b) 用于空间 ATAC-seq 作图的感兴趣区域中 7-AAD 核染色的荧光图像。
(c,d) 无监督聚类分析(c)和小鼠大脑中每个簇的空间分布(d)。为了更好的可视化,我们缩放了像素的大小。
(e) 不同簇中所选标记基因的基因得分的空间映射。
(f,g) 小鼠大脑 scATAC-seq 的整合 (f) 和空间 ATAC-seq (g)
(h) 通过从 scRNA-seq 到空间 ATAC-seq 的标签转移鉴定的选定细胞类型的空间映射。
(i) 包含单个核的像素的空间位置。
(j) 包含单个核的选定像素的荧光图像。
(k) 包含单个核的选定像素的基因分数的热图。
# Figure 4: Spatial chromatin accessibility mapping of a human tonsil with a 20µm pixel size.
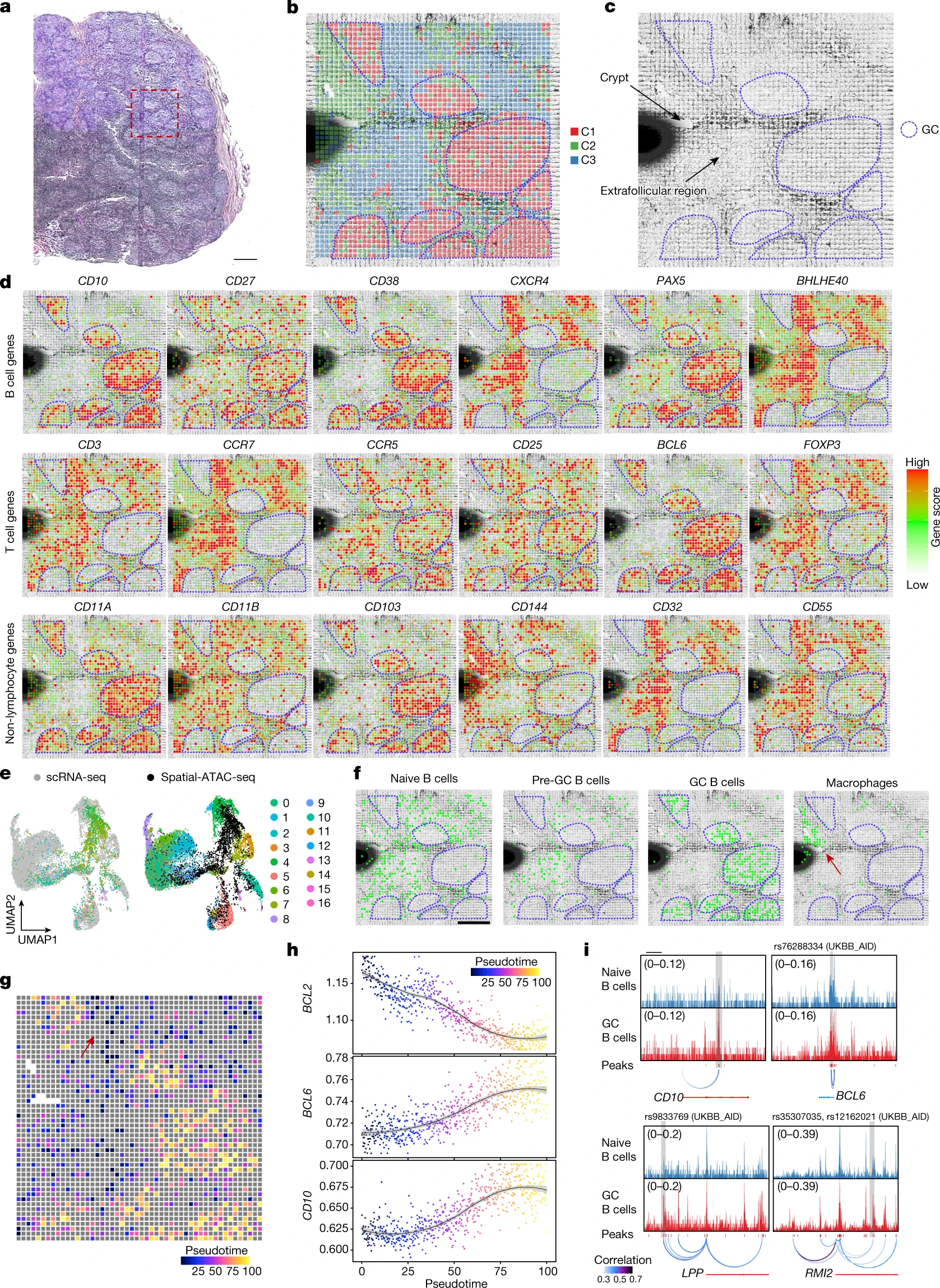
Figure 4. 像素大小为 20 µm 的人类扁桃体的空间染色质可及性图
(a) 来自邻近组织切片的人类扁桃体的 H&E 图像和用于空间染色质可及性映射的感兴趣区域。
(b) 无监督聚类分析和每个聚类的空间分布。为了更好的可视化,我们缩放了像素的大小。
(c) 主要扁桃体区域的解剖注释。
(d) 所选基因的基因得分的空间映射。
(e) scRNA-seq 数据和空间-ATAC-seq 数据的整合。组合数据的无监督聚类按不同的细胞类型着色。
(f) 通过从 scRNA-seq 转移到空间 ATAC-seq 数据的标签来识别选定细胞类型的空间映射。
(g) 在空间中绘制的从初始 B 细胞到 GC B 细胞的发育过程的伪时间重建。
(h) 所选基因的基因分数沿g中所示的伪时间的动态。
(i) 各个调控元件的染色质可及性沿伪时间的动态(在灰色框中突出显示)。显示了精细定位的自身免疫相关 GWAS 变异和定位于可及染色质的高分辨率个体单核苷酸多态性位点。
# Discussion:
我们开发了空间 ATAC-seq,用于完整组织切片中染色质可及性的空间解析分析,并在细胞水平(20 μm 像素大小)保留空间信息。通过使用免疫荧光成像识别仅包含一个核的像素,也可以在没有组织解离的情况下获得单细胞染色质可及性。Spatial-ATAC-seq 应用于小鼠胚胎(E11 和 E13),以描绘器官发生的表观遗传景观;我们确定了具有不同染色质可及性状态的所有主要组织类型,并揭示了发育中的时空变化。绘制小鼠和人类大脑中可访问的基因组图谱揭示了大脑区域的复杂区域化。我们还使用空间 ATAC-seq 绘制了人类扁桃体中不同免疫细胞的表观遗传状态图,并揭示了 B 细胞激活对 GC 反应的动态以及精细绘制的自身免疫 GWAS 遗传变异的推定靶基因。与 Spatial-CUT&Tag 相比,用于组蛋白修饰的靶向分析如图30所示,空间-ATAC-seq提供了全基因组染色质可及性图谱,为此获得高信噪比具有挑战性,特别是在新鲜冷冻组织切片中。
进一步发展的领域包括以下几个方面。首先,可以通过增加条形码的数量(例如,100×100)或使用组织阵列的蛇形微流体通道来进一步增加映射像素的数量。其次,与单细胞技术不同,空间 ATAC-seq 中的像素可能包含部分细胞核或多个细胞核,并可能导致细胞混合物可能包含多种细胞类型,从而使数据解释复杂化。这一挑战可以通过使用细胞类型反卷积方法或与高分辨率组织图像(即多色免疫荧光图像)无缝集成来识别每个像素中的细胞来解决。我们观察到大量像素 (20 μm) 包含单核,这可以产生空间定义的 scATAC-seq 数据。第三,与转录组学和蛋白质组学等其他空间组学测量相结合,可以提供细胞类型和细胞状态的全面图片。我们可以组合用于 DBiT-seq 的试剂5和空间 ATAC-seq 在相同的微流控条形码步骤中实现空间多组学分析,这在理论上应该有效,但确实需要进一步优化组织固定和反应条件以使这些测定兼容。最后,空间 ATAC-seq 尚未进一步扩展到人类疾病患者的组织样本,以充分发挥其在临床研究中的潜力。我们预计空间 ATAC-seq 将为空间生物学增添一个新的维度,从而能够以空间解析的方式分析调控元件,这是空间转录组学或蛋白质组学无法实现的。Spatial-ATAC-seq 可能会改变多个生物医学研究领域,包括发育生物学、神经科学、免疫学、肿瘤学和临床病理学。
