 Motif-based models accurately predict cell type-specific distal regulatory elements
Motif-based models accurately predict cell type-specific distal regulatory elements
题目: 基于模体的模型可准确预测细胞类型特异性的远端调控元件
DOI: https://doi.org/10.1038/s41467-025-65362-2 (opens new window)
Cite: Cornejo-Páramo, P., Zhang, X., Louis, L. et al. Motif-based models accurately predict cell type-specific distal regulatory elements. Nat Commun 16, 10370 (2025).
作者介绍:
| Emily S. Wong |
|---|
 |
| Victor Chang Cardiac Research Institute, Darlinghurst, NSW, Australia |
| e.wong@victorchang.edu.au |
# Abstract:
解析 DNA 序列如何决定细胞类型特异性的调控活性,是基因调控领域中的一个核心挑战。我们提出了一种计算框架 Bag-of-Motifs(BOM),将远端顺式调控元件表示为转录因子(TF) motif 的无序计数集合。这种极简的表示方式结合梯度提升树模型,使得在小鼠、人类、斑马鱼和拟南芥等多种数据集中,能够准确预测细胞类型特异性的增强子。
尽管方法非常简洁,BOM 在使用更少参数的情况下,性能却优于更复杂的深度学习模型。我们还通过实验验证了 BOM 的预测结果:利用最具预测力的 motif 组合构建合成增强子,结果表明这些 motif 集合能够驱动细胞类型特异性的基因表达。
通过提供直接的可解释性和广泛的适用性,BOM 揭示了远端调控区域中一种高度可预测的序列编码规则,并为在不同物种和条件下解析顺式调控语法提供了一个可扩展的计算框架。
# Main:
# Figure 1: BOM accurately classifies mouse embryonic context-specific CREs..
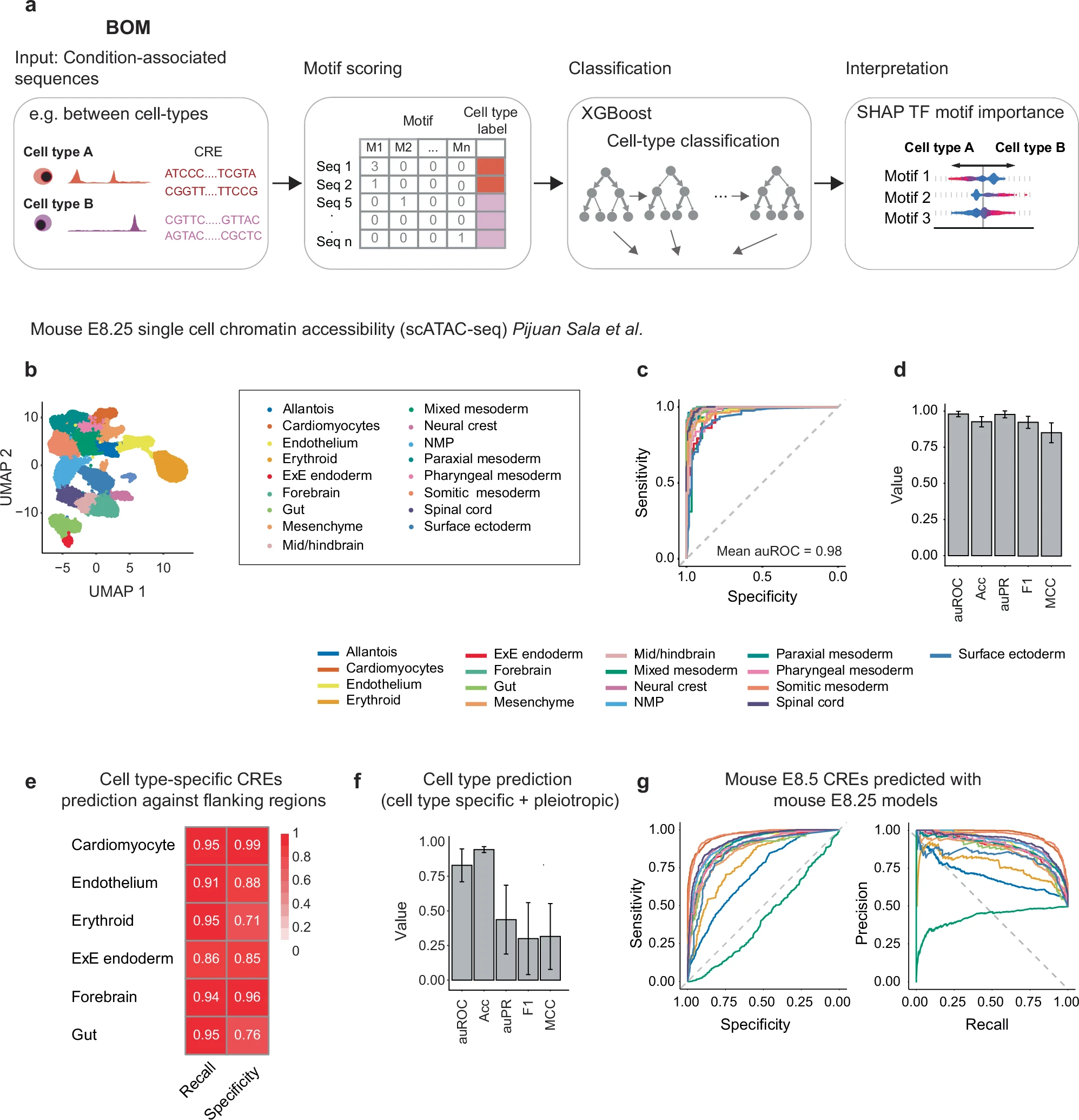
Figure 1. BOM 准确地区分小鼠胚胎中具有特定发育背景的 CRE
(a) BOM 的整体框架。 首先,我们定义至少两组分别特异于不同细胞类型或条件的 CRE 序列。随后,在这些序列中识别转录因子(TF)结合 motif 实例。采用 GimmeMotifs 中的脊椎动物 motif 对 CRE 进行注释。模型使用 XGBoost 进行训练,可完成二分类或多分类的细胞状态判别。通过计算 SHAP 值来解释不同 TF 结合 motif 在分类任务中的重要性。
(b) 参考文献 25 中的小鼠 E8.25 snATAC-seq 数据集概览。
(c) 小鼠 E8.25 胚胎数据集中各细胞类型的二分类 ROC 曲线。
(d) 用于区分 17 种细胞类型特异性 CRE 的二分类 BOM 模型预测性能汇总。
(e) 以 CRE 及其侧翼区域作为负类时,对细胞类型特异性 CRE 进行分类的召回率和特异性。
(f) 在 18 种细胞类型中,用于区分细胞类型特异性 CRE 与多效性(pleiotropic)CRE 的 BOM 模型预测性能汇总。
(g) 使用在小鼠 E8.25 CRE 上训练的模型,对小鼠 E8.5 CRE 进行分类的 ROC 曲线(左)和精确率–召回率曲线(右)。
# Figure 2: BOM outperforms other methods for CRE binary classification..
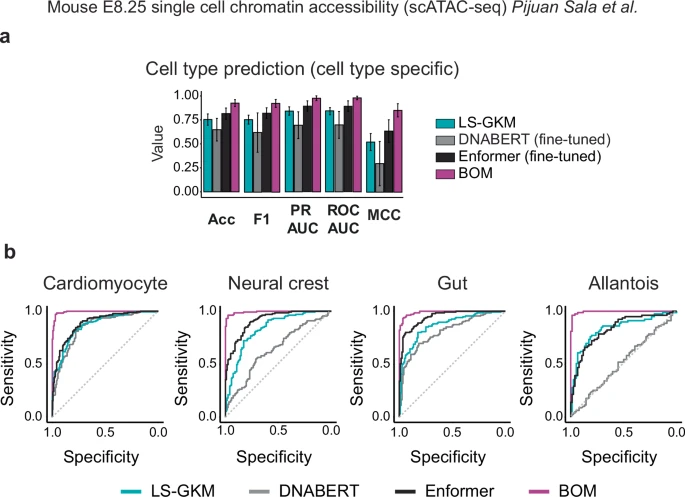
Figure 2. BOM 在 CRE 二分类任务中优于其他方法
(a) 使用 BOM、DNABERT(微调)、Enformer(微调)和 LS-GKM 对 17 种小鼠胚胎细胞类型的 scATAC-seq 峰进行细胞类型特异性的二分类比较。
(b) 四种细胞类型的 ROC 曲线。
# Figure 3: BOM accurately classifies context-specific CREs in different datasets.
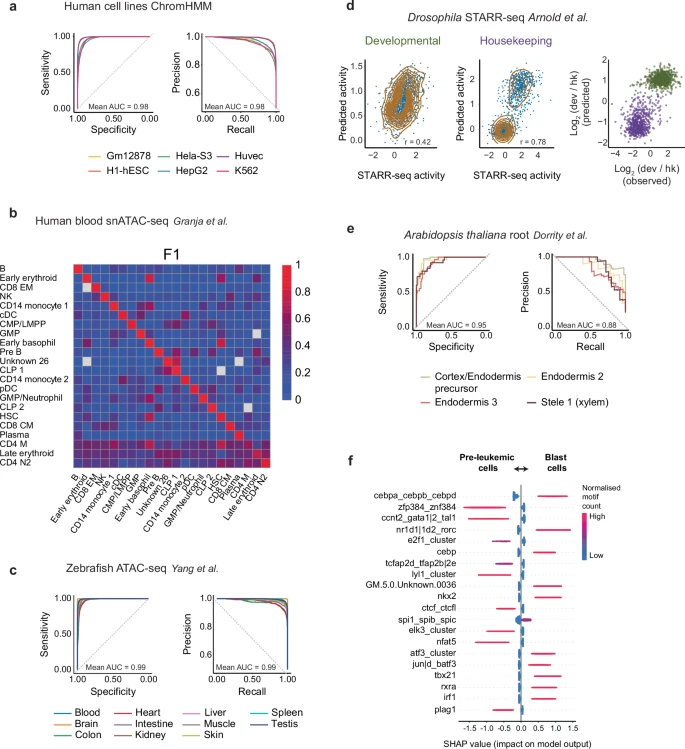
Figure 3. BOM 在不同数据集中准确分类特异性 CREs
(a) ROC 曲线(左)和精确率–召回率曲线(右),展示二分类 BOM 模型在预测六种人类细胞系(Gm12878、H1-hESC、HeLa-S3、HepG2、Huvec、K562)(n = 66,863 CREs)特异性 CRE 的性能。
(b) 二分类 BOM 模型在区分 22 种人类血液和骨髓细胞类型的细胞类型特异性 CRE 上的性能。模型训练目标是从其他细胞类型特异性 CRE 背景中区分目标细胞类型特异性 CRE。
(c) ROC 曲线(左)和精确率–召回率曲线(右),显示基于大体积 ATAC-seq 数据 对 11 种成年斑马鱼组织特异性 CRE 的预测。
(d) 果蝇 S2 细胞中通过 MPRA 测量的发育(Dev)和 housekeeping(Hk)增强子活性与预测活性之间的相关性(左、中面板)(n = 1,258,1,258;Dev 和 Hk 增强子)。右面板显示 Dev 相对于 Hk 增强子的 log2 倍数变化,对比 MPRA 测量值与预测值。增强子颜色根据观察到的类别区分。
(e) 对四种拟南芥根细胞类型40 的细胞类型特异性 CRE 的多分类 BOM 模型分类的 ROC 曲线(左)和 PR 曲线(右)。
(f) 二分类模型中用于区分前白血病细胞或原始 blast cells 中更易接近峰值的 20 个最具预测性的 motif。每个点代表一个 CRE。Y 轴标签为 GimmeMotifs 中的 TF motif,颜色表示归一化的 motif 计数。
# Figure 4: BOM identifies cell-type-specific CREs and cell types across species.
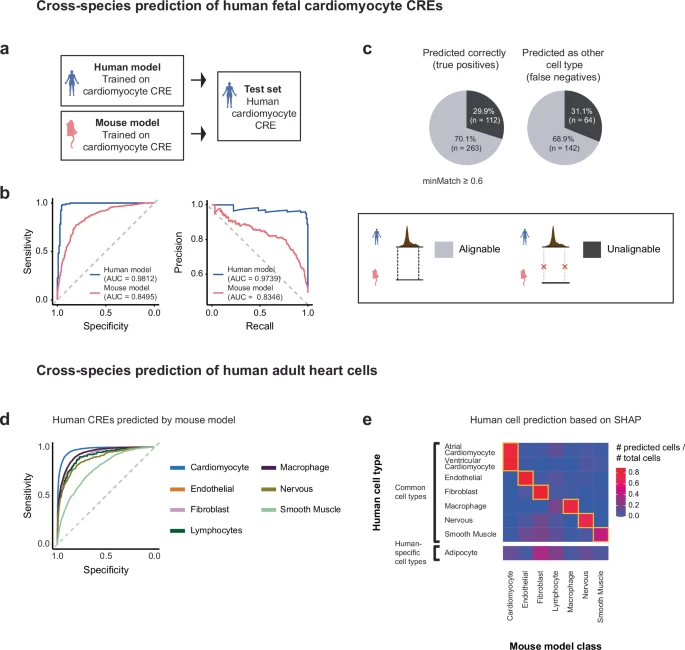
Figure 4. chromVAR从头识别与单细胞染色质可及性变异相关的 motif
(a) 训练二分类模型,用于将人类胎儿心肌细胞特异性的 CRE或小鼠 E8.25 心肌细胞特异性的 CRE与其他细胞类型特异性的 CRE 背景集合区分开来。随后,使用这两种模型对人类 CRE进行预测,该 CRE 集合为训练过程中未使用的独立数据集。
(b) 利用基于人类(蓝色)或小鼠 motif 计数训练的模型,对人类胎儿心肌细胞 CRE进行预测的 ROC 曲线(上)和精确率–召回率曲线(下)。图中标注了每种情况下的曲线下面积(AUC)值。
(c) 可与小鼠基因组比对(灰色)和不可比对(黑色)的人类心肌细胞 CRE 所占比例。CRE 进一步分为预测正确的 CRE(真阳性)和被错误预测为背景集合的 CRE(假阴性)。CRE 通过 liftOver 映射到小鼠基因组,阈值设为 minMatch ≥ 0.6,其中 minMatch 表示需要重新映射的碱基的最小比例。
(d) 使用小鼠多分类模型预测人类成年心脏 CRE的 ROC 曲线。
(e) 人类细胞(n = 55,886)被预测为小鼠多分类模型中各细胞类型的比例。热图上部显示人类与小鼠共有的细胞类型,人类数据集中特有的细胞类型(如脂肪细胞)显示在底部。人类细胞类型与小鼠模型类别之间的对应关系以黄色方框标出。
# Figure 5: BOM provides local and global motif importance scores.
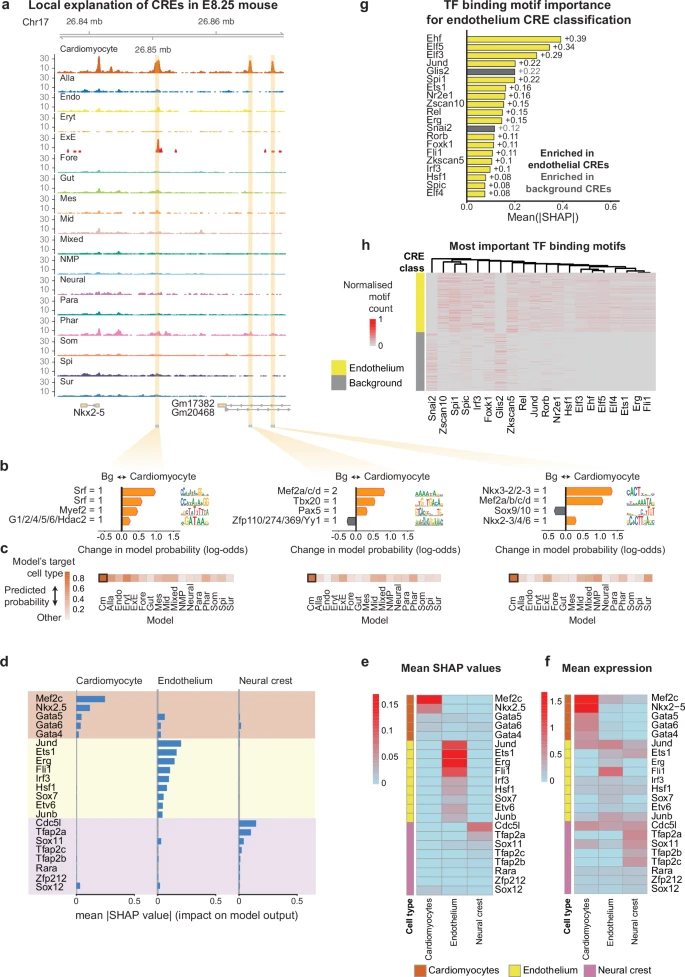
Figure 5. BOM 提供局部和全局的 motif 重要性评分
(a) 基因组浏览器轨迹显示了小鼠 E8.25 各细胞类型在小鼠 17 号染色体 Nkx2-5 基因附近区域的 snATAC-seq 信号。底部标出了 3 个心肌细胞特异性的 CRE位置。
(b) 对 (a) 中展示的 3 个心肌细胞特异性 CRE 的 SHAP 局部解释。图中显示了用于对这些 CRE 进行分类的 最重要的前 4 个 motif 。红色和蓝色箭头表示 SHAP 值的符号(及其方向)。每个 motif 均赋予了一个代表性名称,并标注了对应的 motif 计数。
(c) 热图展示了针对 (a、b) 中 CRE 的预测概率,这些预测来自在小鼠 E8.25 中训练、用于识别各细胞类型特异性 CRE 的 二分类模型。
(d) 在区分小鼠 E8.25 中心肌细胞、内皮细胞和神经嵴相关 CRE 时,顶级转录因子(TF)结合 motif 的平均 |SHAP 值|。SHAP 值基于其他细胞类型特异性 CRE 的集合计算得到。
(e) 展示了 (d) 中 TF 结合 motif 的 平均 SHAP 值。
(f) 与 (d) 和 (e) 中 motif 相对应的 TF 的平均表达水平。表达数据为匹配的 scRNA-seq 实验⁵⁴所得的归一化计数。
(g) 区分小鼠 E8.25 内皮细胞 CRE 的 最重要的前 20 个 motif 。 motif 按照 绝对 SHAP 值的平均值进行排序。黄色和灰色柱状条分别表示 正向和负向的 SHAP 值。
(h) (g) 中所示 motif 的 归一化 motif 计数。 motif 计数的归一化方式同 (d)。
# Figure 6: BOM identifies motifs that drive cell type-dependent activity.
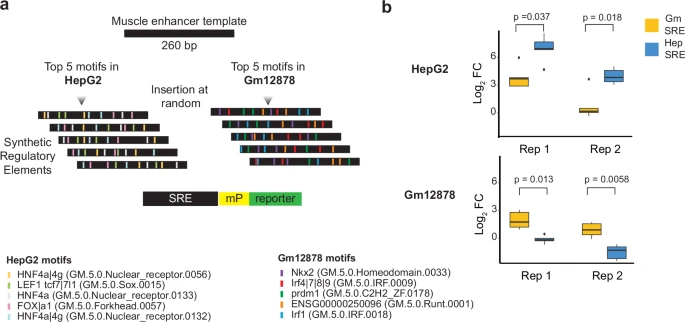
Figure 6. BOM 识别驱动细胞类型依赖性活性的 motif
(a) 将 HepG2 和 GM12878 细胞中 SHAP 排名前 5 的基序分别嵌入到同一条公共模板序列中(每条序列中包含 2 个该基序拷贝),构建合成增强子(SREs)(HepG2 SRE,n = 5;GM12878 SRE,n = 5)。随后,将每个 SRE 插入到含有最小 TATA 启动子的报告载体中,并分别转染到这两种细胞类型中。
(b) 箱线图显示了在校正转染效率和模板增强子活性后,SRE 数量的 log₂ 倍数变化。
# Discussion:
转录因子(TF)结合基序的数量及其组合,在胚胎期和成体脊椎动物中,对细胞状态具有极强的预测能力。
BOM 能够基于与该预测最相关的一组 TF 基序来解释每一条序列,从而帮助界定塑造不同细胞状态的基序集合。
在细胞类型分类任务上,BOM 的表现优于基础深度学习模型 DNABERT 和 Enformer。 我们认为,基于序列的深度学习方法的性能可能受限于可用于训练大量细胞状态的数据规模不足。 基于我们的结果,在预训练深度神经网络(DNN)之上进行迁移学习,是构建远端调控贡献准确表征的一条有前景的路径。
