 SODB facilitates comprehensive exploration of spatial omics data
SODB facilitates comprehensive exploration of spatial omics data
题目: SODB 促进空间组学数据的全面探索
DOI: https://doi.org/10.1038/s41592-023-01773-7 (opens new window)
Cite:Yuan, Z., Pan, W., Zhao, X. et al. Publisher Correction: SODB facilitates comprehensive exploration of spatial omics data. Nat Methods 20, 623 (2023).\
作者介绍:
| Michael Q. Zhang |
|---|
 |
| The University of Texas, Richardson, TX, USA |
| michael.zhang@utdallas.edu |
# Abstract:
空间组学技术产生丰富但高度复杂的数据集。在这里,我们介绍空间组学数据库(SODB),一个基于网络的平台,提供丰富的数据资源和一套交互式数据分析模块。SODB 目前维护着来自超过 2,400 个实验的超过 25 种空间组学技术,这些实验可以作为与各种计算包兼容的统一数据格式免费访问。SODB还提供了多个交互式数据分析模块,特别是一个独特的模块,空间组学视图(SOView)。我们进行全面的统计分析,并使用多个空间组学数据集说明基本和高级分析模块的实用性。我们通过大脑空间转录组数据展示了 SOView 的实用性,并恢复了已知的解剖结构。 我们进一步描绘了具有相关标记基因的功能组织域,这些标记基因在使用以前的方法进行分析时被掩盖。我们最终展示了 SODB 如何有效地促进计算方法的开发。SODB 网站是 https://gene.ai.tencent.com/SpatialOmics/ (opens new window)。命令行包可从https://pysodb.readthedocs.io/en/latest/ (opens new window)获取。
# Results:
# Figure 1: Overview.

Figure 1. 概述
(a – f) SODB的总体设计。六个六边形概括了SODB的六大特点。SODB包含各种类型的空间组学数据(a),这些数据被处理并呈现为统一的Anndata格式(b)。SODB支持交互式探索(e)和定制的数据统计模块(f)。SODB还提供了一个独特的数据探索模块,名为空间组学视图(SOView)。SOView同时支持高效的数据可视化(d)和交互式数据分析(c)功能。六个六边形特征分为三组:数据管理(a,b),基础探索(e,f)和高级探索(c,d)。对于每个组,更详细的信息以不同颜色的标题显示。
(b) 不同空间技术的信息,包括轮廓分子的类型、分子特征的数量和空间分辨率。并对不同平台所包含的空间技术进行了比较:SODB、STomicsDB、SpatialDB 和 SOAR。
# Figure 2: Data characteristics and statistics.
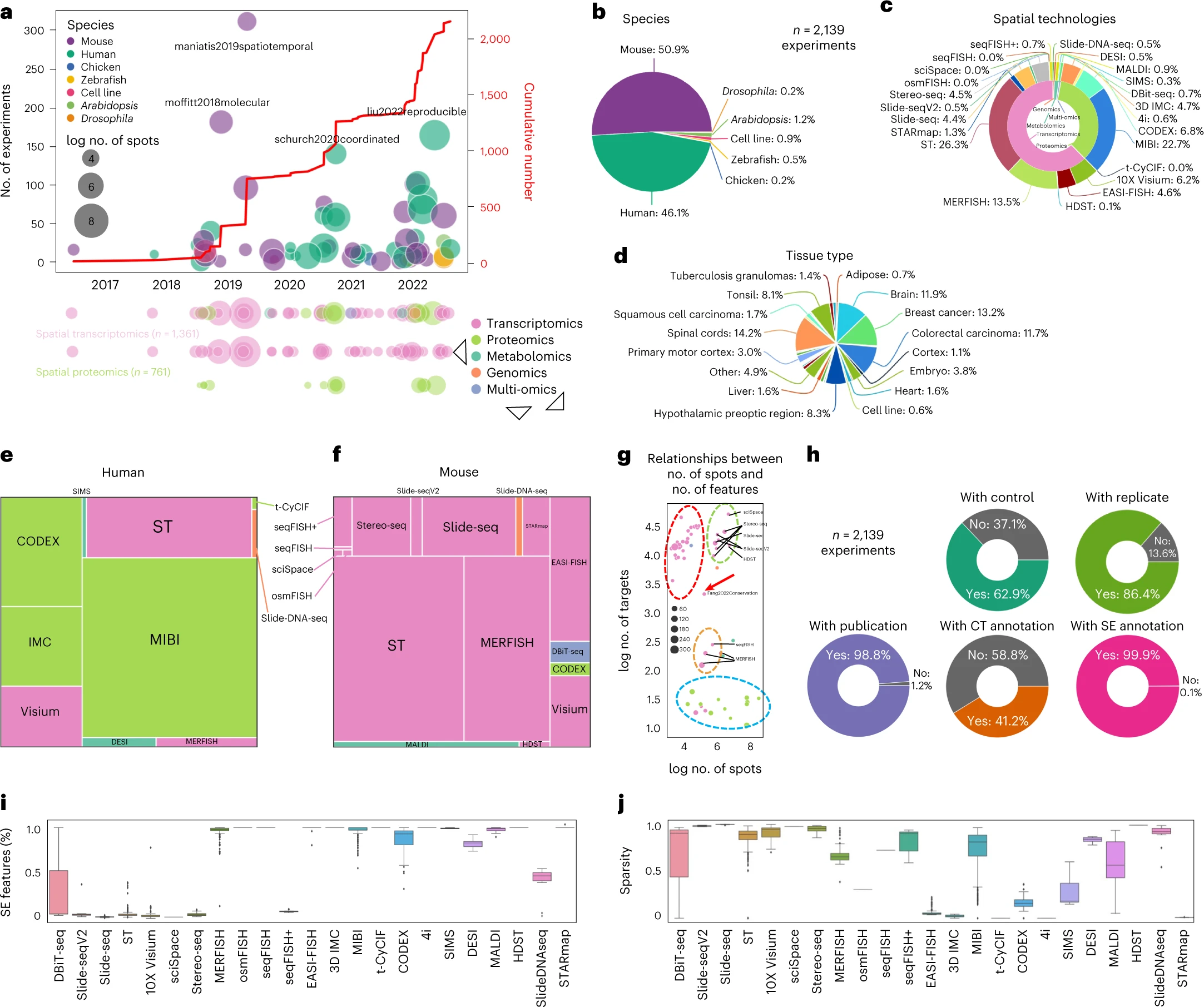
Figure 2. 数据特征和统计
(a) 散点图显示数据集的生成日期,每个点都是一个数据集,按物种着色,按点数量调整大小,y轴是数据集中的实验数量,x轴是发布时间数据集的。线图显示随时间推移的累积实验次数。时间线图显示了不同类别空间技术随时间的实验数量。
(b–d) 饼图显示按物种 ( b )、技术 ( c ) 和组织类型 ( d ) 划分的实验分布。
(e,f) 显示人类 ( e ) 和小鼠 ( f ) 研究中使用的技术分布的树状图。
(g) 散点图显示 spot 数量和分子特征数量之间的关系。每个点都是一个数据集,根据空间转录组学技术的类别着色,并根据实验数量确定大小。不同颜色的虚线圆圈是不同的簇。黑色实线表示数据集的空间技术。红色箭头表示具有代表性的 MERFISH 数据集。
(h) 饼图显示不同质量控制的实验分布。
(i) 箱线图显示按不同技术分组的 SE 功能的百分比。
(j) 箱线图显示按不同技术分组的表达矩阵的稀疏性。
# Figure 3: Interactive views of SODB.
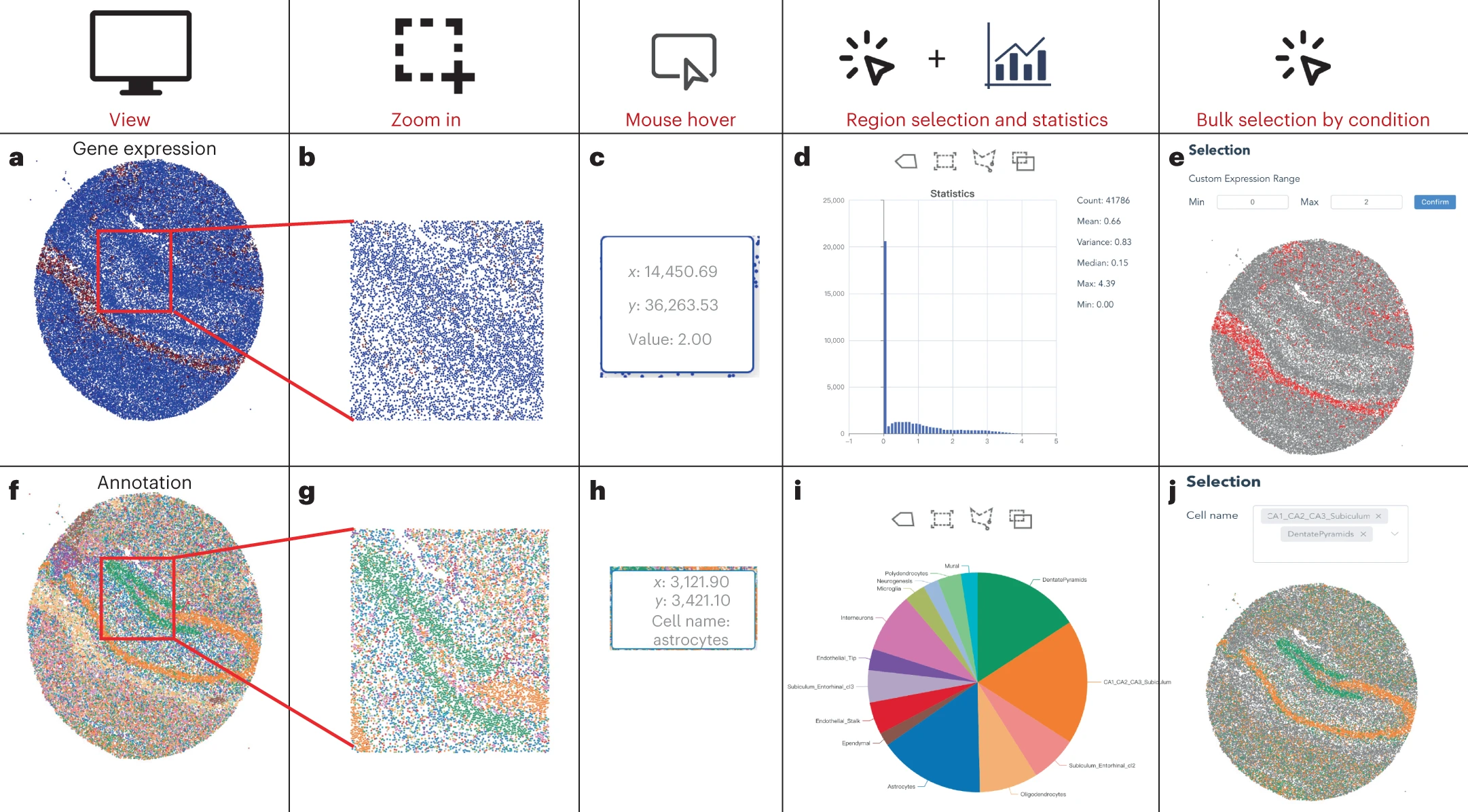
Figure 3. SODB 的交互式视图
(a,f) 交互式数据探索有四种视图:表达式视图(a),注释视图(f),比较视图(补充图10)和SOView(图4及补充图)。
(a–e) 在表达式视图(a)中,可以放大以查看ROI的详细空间表达式(b),鼠标悬停以查看感兴趣斑点的空间和表达信息(c),获取所选ROI内基因表达的分布信息(d),并检查条件(e)选择的斑点的空间分布。
(f–j) 在注释视图(f)中,可以放大以查看ROI内的详细空间像元类型分布(g),鼠标悬停可查看感兴趣点的空间和像元类型信息(h),获取所选ROI内像元类型的分布信息(i)并查看所选像元类型的空间分布(j)。
# Figure 4: SOView demonstration of various spatial omics datasets.
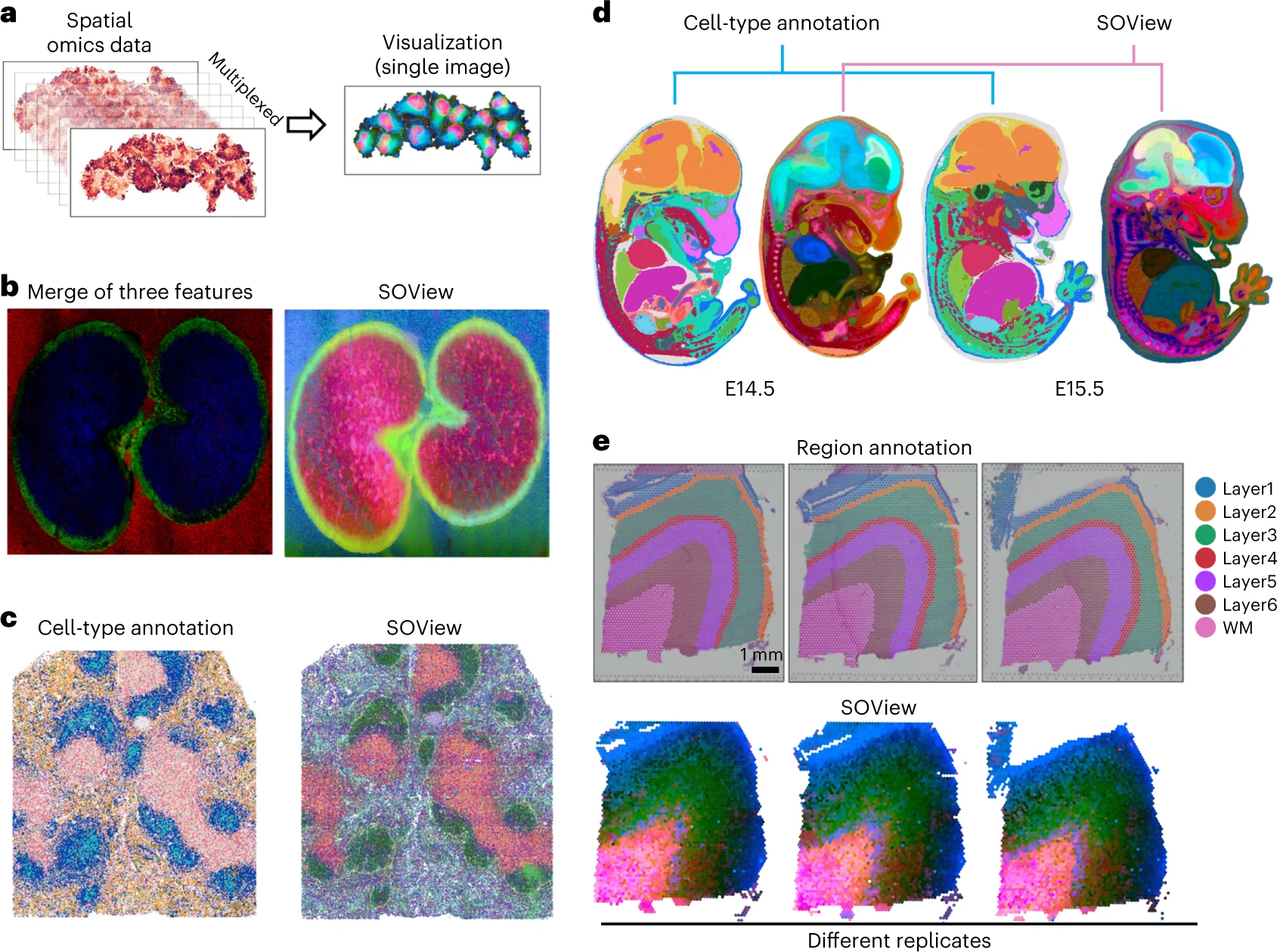
Figure 4. 各种空间组学数据集的SOView演示
(a) SOView输入空间组学数据并输出单个彩色图像以进行交互式可视化。
(b) 空间代谢组学小麦种子数据集上三个单独离子图像(左)和SOView(右)合并之间的可视化比较。
(c) 空间蛋白质组小鼠脾数据集上细胞类型映射(左)和 SOView(右)之间的可视化比较。
(d) 空间转录组学小鼠胚胎数据集上细胞类型映射(左)和SOView(右)之间的可视化比较。
(e) 空间转录组学人类皮层数据集上区域注释(顶部)和 SOView(底部)之间的可视化比较。
# Figure 5: SOView discovers both known and unknown functional tissue domains.
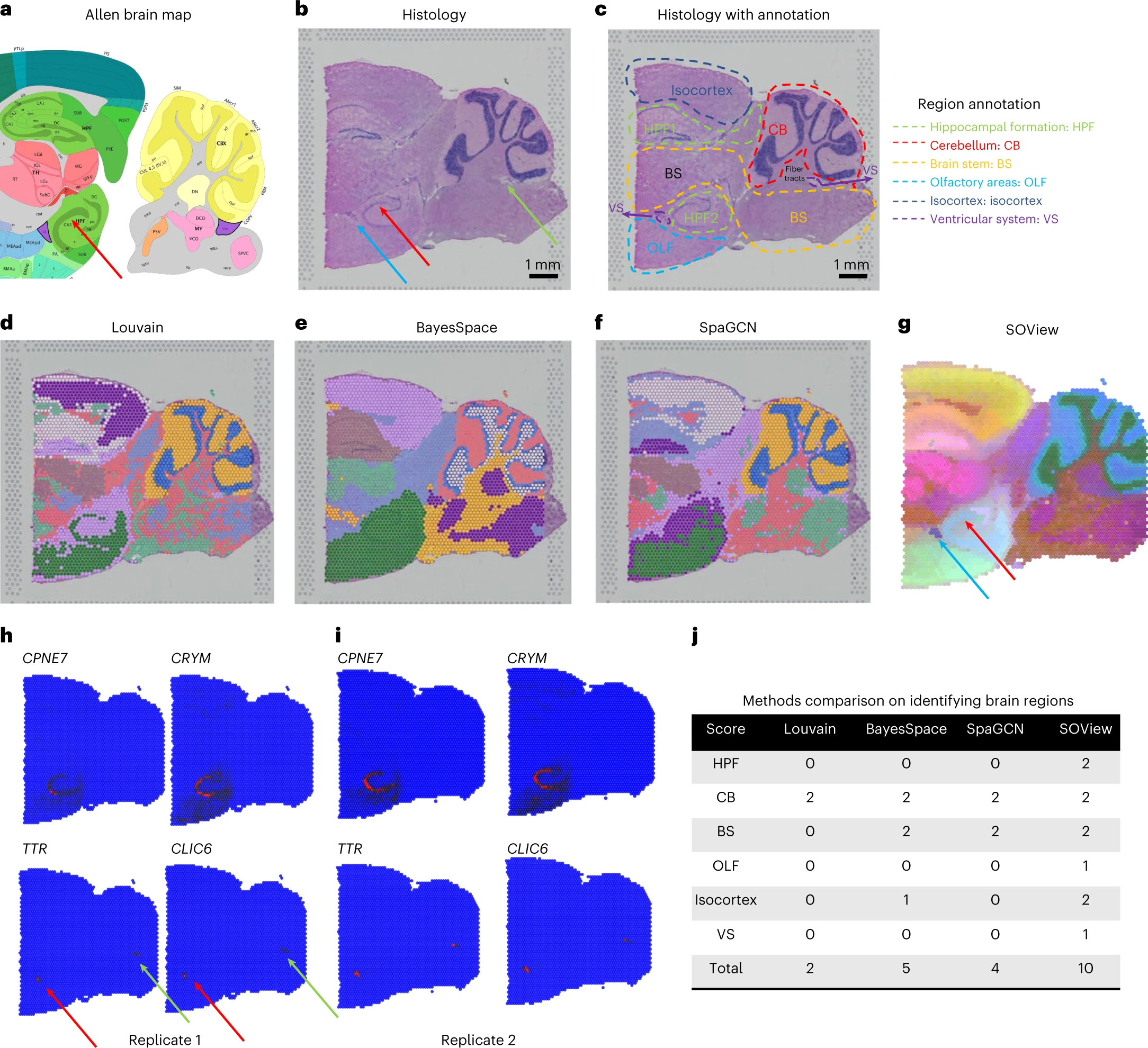
Figure 5. SOView 发现已知和未知的功能组织域
(a,b) 艾伦脑图参考(a)和小鼠大脑后部的组织学图像( b )。
(c) 主要大脑区域参考a和补充图20进行注释。
(d–g) 不同方法描绘不同大脑结构的结果:Louvain ( d )、BayesSpace ( e )、SpaGCN ( f ) 和 SOView ( g )。
(h) 顶部,CA3sp 区域的顶部标记基因,在 SOView ( g ) 红色箭头中识别。最重要的标记基因是通过SODB获得的(补充图19)。VS 区域的底部、顶部标记基因,在 SOView ( g ) 蓝色箭头中识别。最重要的标记基因是通过SODB获得的
(i) 与h中相同的基因在另一个重复中显示出一致的模式。
(j) 不同方法区分不同大脑区域能力的比较。
# Figure 6: SOView demonstration of various spatial omics datasets.
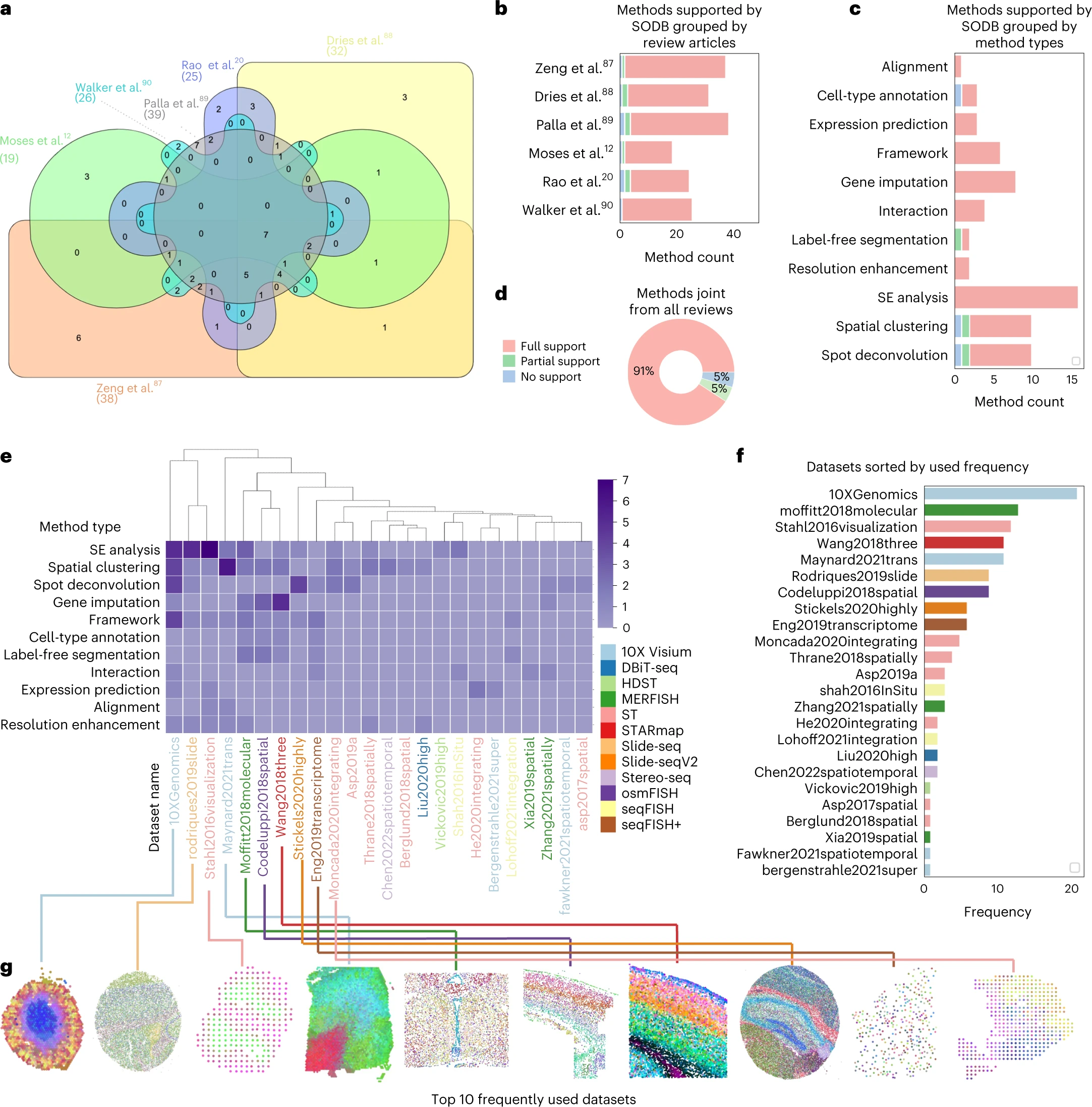
Figure 6. 各种空间组学数据集的SOView演示
(a) 维恩图显示了六篇高影响力评论文章中提到的计算方法。
(b,c) 条形图显示SODB 完全/部分/不支持多少种计算方法(按评论文章 ( b ) 或方法类型 ( c ) 分组)。
(d) 饼图显示SODB 完全/部分/不支持多少种方法。
(e) 热图显示每种方法类型使用的每个数据集的频率。数据集名称由空间技术着色。
(f) 条形图显示最常用的数据集。
(g) SOView 十大常用数据集的可视化。在每个数据集中,使用一个实验进行可视化。
# Discussion:
我们推出了 SODB,一个基于网络的平台,结合了大规模数据沉积和通用空间组学数据的交互式数据探索。SODB提供了各种类型的空间组学数据集(例如,空间转录组学、蛋白质组学、代谢组学、基因组学和多组学),并具有可下载的统一数据格式,可以直接输入到许多主流分析包中。除了数据之外,SODB 还提供了一套交互式数据探索模块。其中,SOView是SODB的一个关键功能,它可以用来可视化全局组织结构,并识别一些微妙但重要的局部或亚组织结构。结合 SOView 和 SODB 的交互界面,可以通过自动生成的标记基因来表征用户定义的 ROI。
